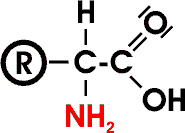
 Der "Rest" (R) oder die "Seitenkette" ist im einfachsten Fall, bei der Aminoethansäure (=Glycin), ein Wasserstoffatom, sonst eine aliphatische oder aromatische Gruppe.
Der "Rest" (R) oder die "Seitenkette" ist im einfachsten Fall, bei der Aminoethansäure (=Glycin), ein Wasserstoffatom, sonst eine aliphatische oder aromatische Gruppe.| Bio-Ecke | Chemie-Ecke | Home | Unterricht | sOWLrebel |
|---|
Im Zellplasma aller Organismen finden sich makromolekulare Substanzen, die in ihrem Bau und in ihren Eigenschaften dem Eiweiß des Vogeleies gleichen und daher Eiweißstoffe genannt werden. Die von J. Berzelius vorgeschlagene Bezeichnung Protein (proteios [gr.]:der Erste, der Wichtigste) weist auf ihre überragende Bedeutung für das Leben hin. Wie die Kohlenhydrate übernehmen die Proteine eine Vielzahl von Aufgaben innerhalb des Organismus.
Alle Proteine lassen sich durch Erhitzen mit Säuren hydrolytisch in Aminosäuren (Aminocarbonsäuren) spalten. Proteine sind daher als Kondensationsprodukte von Aminosäuren aufzufassen.
Aminosäuren sind auch in freier Form im gesamten Zellbereich weit verbreitet.
 Der "Rest" (R) oder die "Seitenkette" ist im einfachsten Fall, bei der Aminoethansäure (=Glycin), ein Wasserstoffatom, sonst eine aliphatische oder aromatische Gruppe.
Der "Rest" (R) oder die "Seitenkette" ist im einfachsten Fall, bei der Aminoethansäure (=Glycin), ein Wasserstoffatom, sonst eine aliphatische oder aromatische Gruppe.
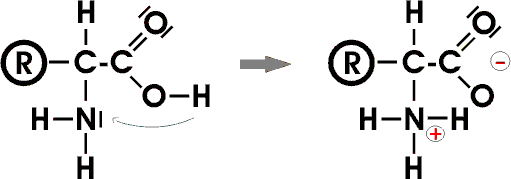
Als "innere Salze" sind die ASen daher in manchen Eigenschaften den Ionenverbindungen ähnlich: Sie sind meist 1) gut wasserlöslich, besitzen 2) hohe Schmelzpunkte und lassen sich 3) nicht unzersetzt destillieren.
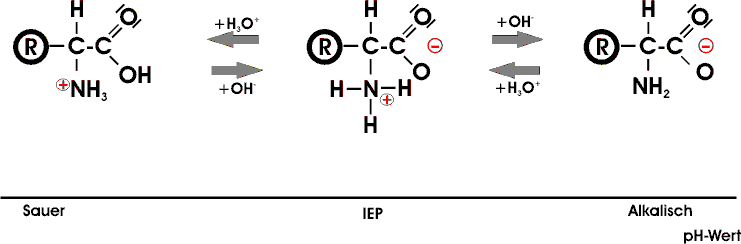
Prinzipiell gleichartig verhalten sich AS, die in ihrem Rest eine zusätzliche saure oder basische Gruppe enthalten. Nur liegt bei der Asparaginsäure mit einer zusätzlichen Carboxylgruppe der IEP im sauren und bei der basischen AS Lysin mit einer zusätzlichen Aminogruppe im basischen Bereich.
Auch Prolin besitzt eine aliphatische Seitenkette; es unterscheidet sich jedoch von
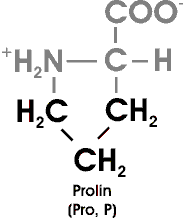
den anderen 19 Aminosäuren dadurch, dass seine Seitenkette sowohl mit dem Stickstoffatom als auch mit dem alpha-Kohlenstoffatom verbunden ist. Die dadurch entstehende Ringstruktur übt einen deutlichen Einfluß auf die Architektur von Proteinen aus. Prolin bricht alpha-Helices ab. Es scheut den Kontakt mit Wasser nicht (die anderen aliphatischen AS sind hydrophob) und ist häufig in den Knicken gefalteter Proteinketten zu finden. Man sollte beachten, dass Prolin eine sekundäre Aminogruppe besitzt und eigentlich richtigerweise als Iminosäure bezeichnet werden müßte.
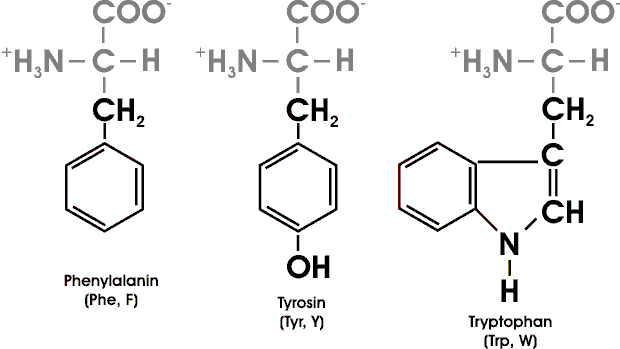
Die aromatischen Ringe von Phenylalanin, Tryptophan und Tyrosin besitzen delokalisierte pi-Elektronenwolken, die es ihnen ermöglichen, mit anderen pi-Systemen in Wechselwirkung zu treten und Elektronen zu übertragen.
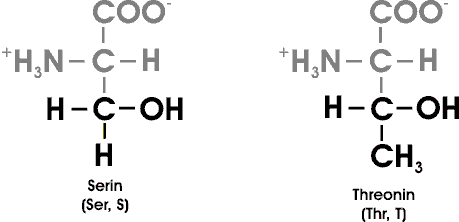
Threonin enthält wie Isoleucin zwei Asymetriezentren. Alle übrigen AS (von den 20), mit Ausnahme des Glycins, besitzen genau ein Asymetriezentrum (der alpha-Kohlenstoff); Glycin ist die einzige optisch inaktive AS.
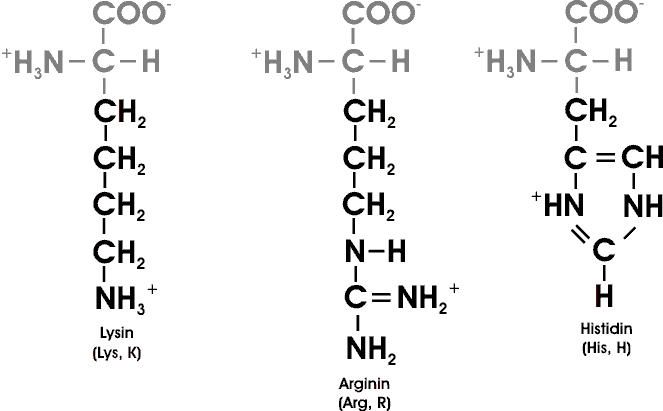
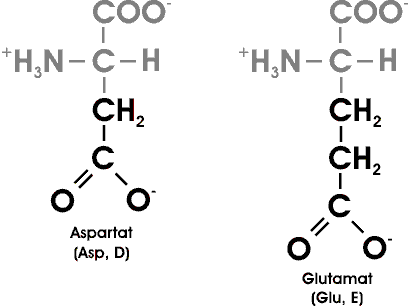
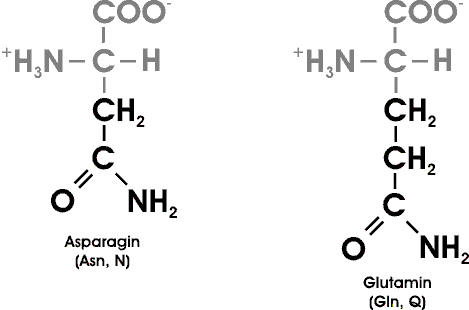
Sieben der 20 AS haben leicht ionisierbare Seitenketten. Noch zwei weitere Gruppen können in Proteinen ionisiert werden: die endständigen Amino- und Carboxyl-Gruppen.
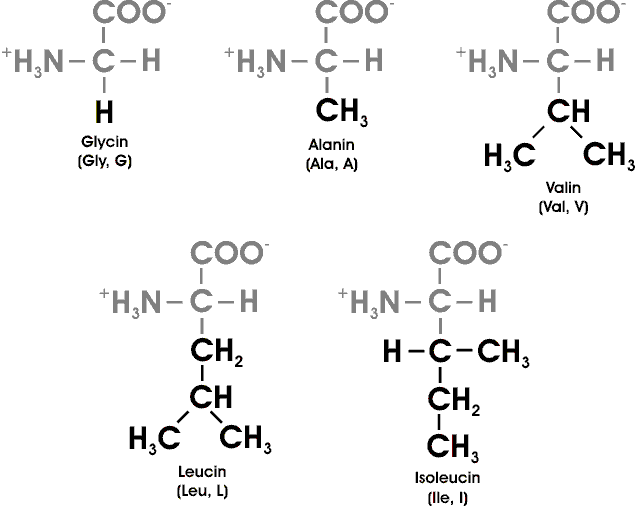
Aminosäuren werden oft mit einer Abkürzung aus drei Buchstaben oder einem Ein-Buchstaben-Symbol bezeichnet. Als Abkürzungen dienen die ersten drei Buchstaben des Namens, ausser bei Tryptophan (Trp), Asparagin (Asn), Glutamin (Gln) und Isoleucin (Ile). Als Symbole hat man für die kleinen AS den Anfangsbuchstaben genommen (etwa G für Glycin); die übrigen Symbole entsprechen einer Konvention. Die Abkürzungen und Symbole sind ein wichtiger Bestandteil des Biochemikervokabulars.
Die aus natürlichen Proteinen gewonnen AS gehören (fast) ausschließlich der L-Reihe an, stimmen in ihrer Konfiguration mit dem linksdrehenden L-Glycerinaldehyd überein. Über die Gründe für die Bevorzugung der L-Konfiguration lassen sich bislang nur Vermutungen anstellen. Vielleicht ist sie eine Folge der Oberflächenstruktur jener Mineralstoffe, auf deren Oberfläche sich die Kondensation der ersten Proteine abspielte. Die Übereinstimmung stützt die Vorstellung, dass alle heute existierende Lebewesen aus einer gemeinsamen "Wurzel" hervorgegangen sind.
Biologisch bedeutsam ist, dass der menschliche Körper nicht alle von ihm benötigten AS selbst herstellen kann. Darauf beruht die Unterscheidung in essentielle AS, die in den Proteinen der Nahrung enthalten sein müssen, und in nichtessentielle AS.
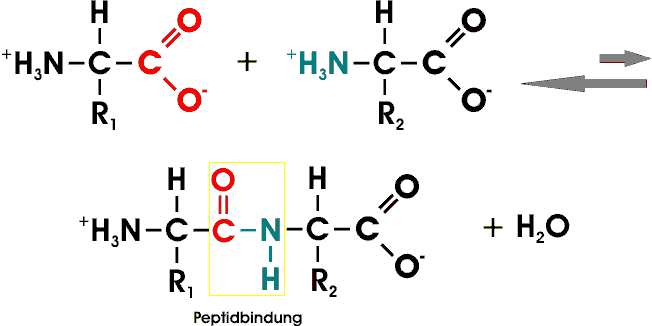
Das Gleichgewicht dieser Reaktion liegt auf seiten der Hydrolyse (Edukte); daher bedarf die Biosynthese von Peptidbindungen eines beträchtlichen Energieaufwands, während ihre Spaltung thermodynamisch feiwillig abläuft.
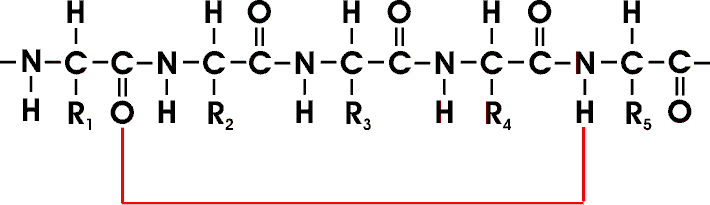
Viele durch Peptidbindung verknüpfte AS bilden eine unverzweigte Polypeptidkette, deren einzelne Aminosäureeinheiten als Reste bezeichnet werden.
Ein Beispiel bietet hier ein Pentapeptid; die einzelnen AS sind eingerahmt. Der Kettenanfang ist am Aminoende (N-Terminus), das Kettenende ist am Carboxyende (C-Terminus). Die Reste können unterschiedlich, aber auch gleich sein.
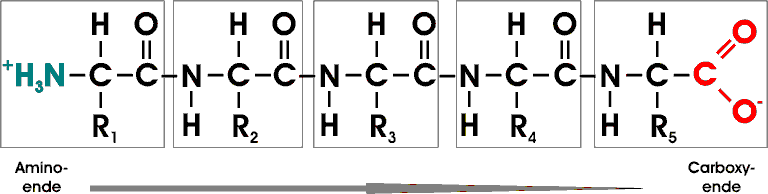
Ein Polypeptid besteht aus sich regelmäßig wiederholenden Einheiten, die die Hauptkette bilden, und einem variablen Anteil, den einzelnen Seitenketten (R).
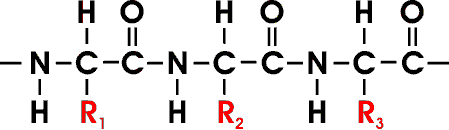
Die Hauptkette wird auch als Rückgrat bezeichnet. Durch die 20 verschiedenen Reste , ist eine hohe Variabilität möglich. Bei einer Kettenlänge von 100 AS ergeben sich 20100 Möglichkeiten der unterschiedlichen Verknüpfung, dies entspricht einer Zahl von 10130. Die meisten natürlich vorkommenden Polypeptidketten enthalten zwischen 50 und 2000 AS. Das mittlere Molekulargewicht einer AS beträgt etwa 110; somit liegt das Molekulargewicht der meisten Polypeptidketten zwischen 5500 und 220 000.
Die Masse eines Proteins wird in Dalton angegeben; ein Dalton (d) entspricht einer Atommasseneinheit. Ein Protein mit einem Molekulargewicht von 50.000 hat also die Masse 50.000 d beziehungsweise 50 kd.
Dalton (d) ist eine Masseneinheit, die der Masse des Wasserstoffatoms (exakt 1 auf der Atommassenskala) sehr nahe kommt. Sie ist nach John Dalton (1766 - 1844) benannt, der die Atomtheorie der Materie entwickelte. Ein Kilodalton (kd) entspricht 1000 Dalton (d).
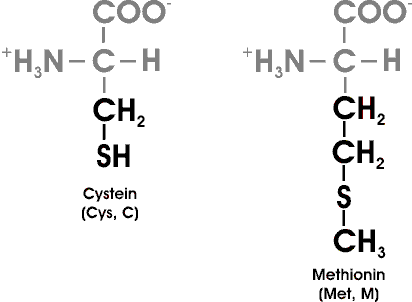
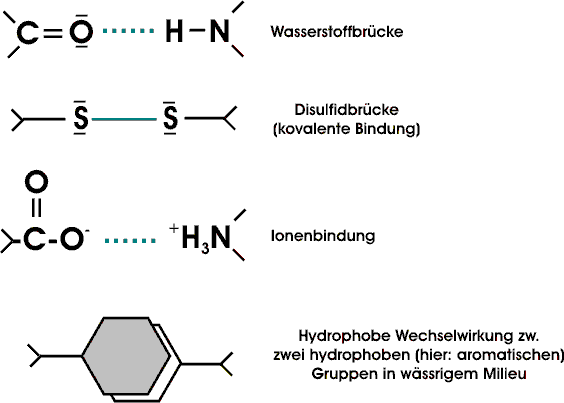
Einige Proteine enthalten Disulfidbindungen; echte kovalente Bindungen. Diese Querverbindungen zwischen Ketten oder Teilen einer Kette enstehen durch Oxidation von Cysteinresten. Das dabei gebildete Disulfid nennt man Cystin.
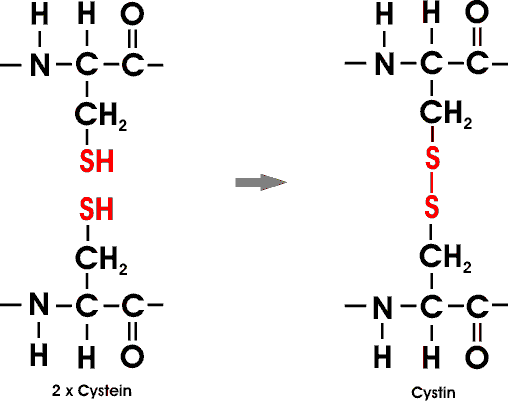
Intrazelluläre Proteine haben meist keine Disulfidbrücken, extrazelluläre dagegen häufig mehrere. In einigen Proteinen findet man Querverbindungen ohne Schwefel, die von Lysinseitenketten abgeleitet sind. Zum Beispiel werden die Kollagenfasern im Bindegewebe sowie Fibrin-Gerinnsel auf diese Weise stabilisiert.
Eine wichtige Vorraussetzung für die dreidimensionale Struktur der Proteine ist die in sich starre und planare Peptidbindung.
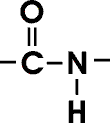 Der Wasserstoff der substituierten Aminogruppe liegt fast immer in trans Position zum Sauerstoff der Carbonylgruppe. Eine freie Rotation um die Bindung zwischen dem Carbonyl-C-Atom und dem Stickstoffatom der Peptidbindung ist nicht möglich, weil diese Bindung partiellen Doppelbindungscharakter besitzt. Die Länge dieser Bindung beträgt charakteristischerweise 0,132 nm, ein Wert, der zwischen dem der C-N-Einfachbindung (0,149 nm) und dem der C=N-Doppelbindung (0,127 nm) liegt. Im Gegensatz dazu ist die Bindung zwischen dem alpha-C-Atom und dem Carbonylkohlenstoffatom eine reine Einfachbindung, genau wie jene zwischen dem alpha-C-Atom und dem Stickstoffatom. Die Bindungen beiderseits der starren Peptidbindung weisen folglich eine große Rotationsfreiheit.
Der Wasserstoff der substituierten Aminogruppe liegt fast immer in trans Position zum Sauerstoff der Carbonylgruppe. Eine freie Rotation um die Bindung zwischen dem Carbonyl-C-Atom und dem Stickstoffatom der Peptidbindung ist nicht möglich, weil diese Bindung partiellen Doppelbindungscharakter besitzt. Die Länge dieser Bindung beträgt charakteristischerweise 0,132 nm, ein Wert, der zwischen dem der C-N-Einfachbindung (0,149 nm) und dem der C=N-Doppelbindung (0,127 nm) liegt. Im Gegensatz dazu ist die Bindung zwischen dem alpha-C-Atom und dem Carbonylkohlenstoffatom eine reine Einfachbindung, genau wie jene zwischen dem alpha-C-Atom und dem Stickstoffatom. Die Bindungen beiderseits der starren Peptidbindung weisen folglich eine große Rotationsfreiheit.
Die alpha-Helix ist eine stabförmige Struktur; die eng aufgewickelte Polypeptidhauptkette bildet den inneren Teil des Stabes, während die Seitenketten in schraubenartiger Anordnung nach aussen weisen.
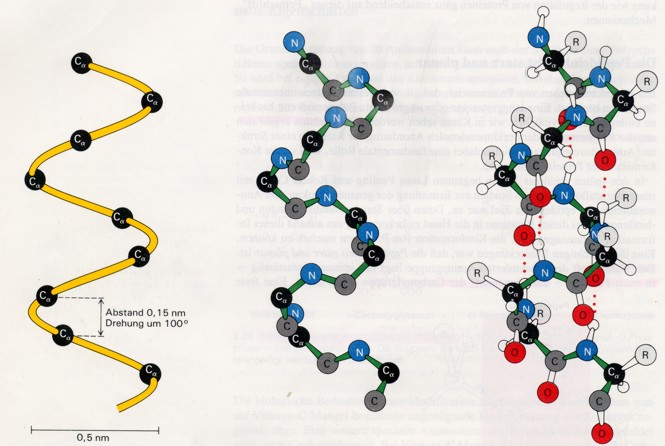
Im Modell sieht man eine rechtsgängige alpha-Helix. Links sieht man nur die alpha-Kohlenstoffatome, in der Mitte ist nur das Rückgrat dargestellt (-N-Calpha-C-), rechts ist die gesamte Helix aufgezeigt.
Die alpha-Helix wird durch Wasserstoffbrücken zwischen den NH- und CO-Gruppen der Hauptkette stabilisiert; dabei bildet die CO-Gruppe jeder AS eine Wasserstoffbrücke zur NH-Gruppe jener AS, die in der linearen Sequenz vier Reste vor ihr liegt.
Folglich sind alle CO- und NH-Gruppen der Hauptkette an Wasserstoffbrücken beteiligt. Jeder Rest ist gegen den nächsten um 0,15 nm entlang der Helixachse versetzt und um 100° verdreht, so dass eine volle Umdrehung der Helix 3,6 Aminosäurenreste entspricht. Deshalb liegen AS, die in der linearen Sequenz drei oder vier Reste voneinander entfernt sind, innerhalb der Helix in unmittelbarer räumlicher Nachbarschaft; dagegen befinden sich solche, die in der linearen Sequenz zwei Reste auseinander sind, auf entgegengesetzten Seiten der Helix und können so kaum miteinander in Kontakt treten.
Die Ganghöhe der alpha-Helix beträgt 0,54 nm - das Produkt aus der Versetzung (0,15 nm) und der Anzahl Reste pro Windung (3,6). Der Schraubensinn einer Helix kann theoretisch nach rechts (im Uhrzeigersinn) oder nach links (im Gegenuhzeigersinn) weisen, doch findet man in Proteinen nur rechtsgängige alpha-Helices.
Die beta-Faltblatt-Struktur unterscheidet sich wesentlich von der alpha-Helix, da sie mehr platten- als stabförmige Gestalt besitzt. Die Polypeptidkette eines beta-Faltblattes ist fast völlig gestreckt und nicht eng verknäult wie in der alpha-Helix. Die axiale Entfernung zwischen benachbarten AS beträgt 0,35 nm gegenüber 0,15 nm bei der alpha-Helix.
Ein weiterer Unterschied besteht darin, dass die beta-Faltblattstruktur durch Wasserstoffbrücken zwischen CO- und NH-Gruppen verschiedener Polypeptidstränge stabilisiert werden kann, während dies bei der alpha-Helix von CO- und NH-Gruppen nur derselben Polypeptidkette besorgt wird.
Benachbarte Stränge in einem beta-Faltblatt können dieselbe Richtung aufweisen (parallele beta-Faltblatt-Struktur) oder entgegengesetzt verlaufen (antiparallele beta-Faltblattstruktur); das Seidenfibroin (Strukturprotein der Seide) beispielsweise besteht fast ganz aus antiparallelen beta-Faltblatt-Strukturen.
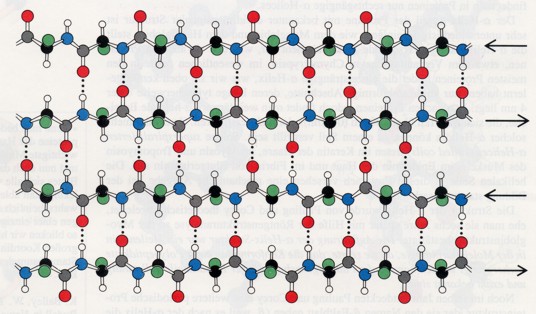
Struktureinheiten aus zwei bis fünf parallelen beta-Strängen treten besonders häufig auf.
Die meisten Proteine haben aufgrund häufiger Richtungsänderungen ihrer Polypeptidketten
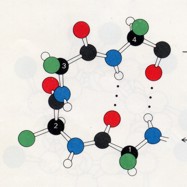 eine kompakte, globuläre Gestalt. Die Untersuchung zahlreicher Proteine hat gezeigt, dass viele Kehren durch ein allgemeines Strukturprinzip, die sogenannte beta-Schleife, zustandekommen.
eine kompakte, globuläre Gestalt. Die Untersuchung zahlreicher Proteine hat gezeigt, dass viele Kehren durch ein allgemeines Strukturprinzip, die sogenannte beta-Schleife, zustandekommen.
Das Besondere bei dieser Schleifenstruktur ist, dass die CO-Gruppe des Restes n eine Wasserstoffbrücke zur NH-Gruppe des Restes (n + 3) ausbildet. So kann eine Polypeptidkette abrupt ihre Richtung umkehren. Beta-Schleifen verbinden häufig antiparallele beta-Faltblatt-Stränge - daher ihr Name. Sie werden auch als Haarnadelschleifen bezeichnet.
Neben Disulfidbrücken, Ionenbindungen zwischen geladenen Resten und Wasserstoffbrücken zwischen polaren Resten treten - in wässriger Lösung - zwischen unpolaren Resten hydrophobe Wechselwirkungen auf. In wässriger Lösung ist eine möglichst kleine Grenzfläche zwischen unpolaren Gruppen und Wasser energetisch günstig. Durch eine Zusammenlagerung mehrerer hydrophober Gruppen wird die Grenzfläche insgesamt verkleinert.
Das Keratin in Haar und Wolle und das Fibroin in der Seidenfaser sind Vertreter der Faserproteine (Skleroproteine). Diese Gerüststoffe sind in Wasser nahezu unlöslich. Dagegen gehen die Kugelproteine (globuläre Proteine) in Wasser und verdünnter Salzlösung kolloidal in Lösung. Beispiele bilden die Globine, wie im Hämoglobin der roten Blutkörperchen und im Myoglobin des Muskels, und viele Enzyme.
Kugelproteine besitzen oft eine recht komplizierte Tertiärstruktur: Beim Myoglobin wechseln starre helicale Abschnitte mit flexibleren Bereichen. Durch die Art der Faltung gelangen die hydrophilen Reste an die Oberfläche und damit in Berührung mit der umgebenden wässrigen Flüssigkeit, während die hydrophoben Reste ins Innere des Kugelproteins eintauchen. Diese Struktur wird durch Bindungen zwischen den Resten stabilisiert.
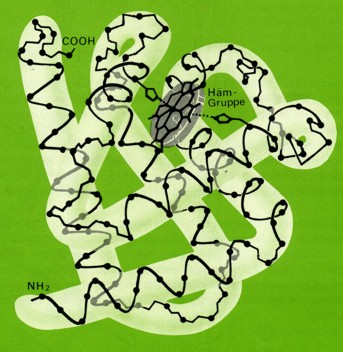 Während viele Proteine ausschließlich aus Aminosäuren aufgebaut sind, ist bei anderen Eiweißstoffen das Protein mit einer andersartigen sog. prosthetische Gruppe verbunden. Man spricht dann von einem zusammengesetzten Protein oder Proteid.
Während viele Proteine ausschließlich aus Aminosäuren aufgebaut sind, ist bei anderen Eiweißstoffen das Protein mit einer andersartigen sog. prosthetische Gruppe verbunden. Man spricht dann von einem zusammengesetzten Protein oder Proteid.Als prosthetische Gruppe besitzen die Chromoproteine einen organische Farbstoff. Die bekanntesten Beispiele sind das Myoglobin, das die Fleischfarbe bedingt, und das Hämoglobin in den roten Blutkörperchen.
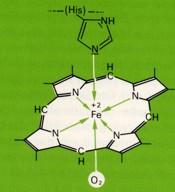 Das Myoglobin ist mit einer Häm-Gruppe verknüpft. Diese enthält einen stickstoffhaltigen Porphyrinring und ein zentrales Eisenatom. Dieses besitzt sechs Koordinationsstellen: Vierfach ist es mit den Stickstoffatomen des Porphyrinringes verbunden. Die fünfte Bindung geht zum Histidinrest des Globins. An die sechste Koordinationsstelle kann zeitweise Sauerstoff angelagert werden.
Das Myoglobin ist mit einer Häm-Gruppe verknüpft. Diese enthält einen stickstoffhaltigen Porphyrinring und ein zentrales Eisenatom. Dieses besitzt sechs Koordinationsstellen: Vierfach ist es mit den Stickstoffatomen des Porphyrinringes verbunden. Die fünfte Bindung geht zum Histidinrest des Globins. An die sechste Koordinationsstelle kann zeitweise Sauerstoff angelagert werden.
Proteine treten mitunter zu noch größeren Einheiten zusammen. Derartige Eiweißkörper bilden eine Quartärstruktur, indem sich einzelne Protein-Ketten zu übergeordneten Strukturen zusammenlagern.
 Hierfür bietet das Hämoglobin ein Beispiel; es besteht aus vier Proteinen, die jeweils dem Myoglobin ähneln und von denen je zwei identisch sind (alpha- und beta-Kette).
Hierfür bietet das Hämoglobin ein Beispiel; es besteht aus vier Proteinen, die jeweils dem Myoglobin ähneln und von denen je zwei identisch sind (alpha- und beta-Kette).
Die künstlich aus dem Hämoglobin isolierten Einzelproteine fügen sich von selbst (self assembling) wieder zusammen. Dies bedeutet, dass die Lage der Kontakstellen zwischen den Proteinen und somit auch die spezifische Quartärstruktur dieses Eiweißstoffes bereits durch die Aminosäurensequenz festgelegt und vorbestimmt ist.
Die Primär-, Sekundär-, Tertiär- sowie die Quartärstruktur wird prinzipiell durch die Aminosäurensequenz des jeweiligen Proteins bedingt.